今天题目里的“重磅”二字略显浮夸,但这个神器完全值得!
近日,国内知名科研工具箱平台科研者之家(HOME for Researchers)发布了首个零基础的分子对接平台——Dockeasy。
这足以重磅,是因为分子对接如果找公司至少千把块,用这个工具,一分钱不要!
科研者之家官网工具位置(还贴心放了视频教程)
为啥这么贵?
因为好看,放文章里显著增加逼格,进而增加期刊接受率(我猜的,但谁不喜欢图片好看的文章呢)。
还因为会做的人少,计算生物学专业的同学会,一般人都不会。
但其实,凡是你的文章或课题结果里涉及到小分子和靶蛋白结合,多肽和蛋白结合,蛋白和蛋白结合的结果,都可以增加分子对接的数据,相当于增加了原子水平的的结合证据,如此显然就可以让结论更加Solid啦。
随便搜一篇,比如在这篇Nature Communications上的文献里,作者在实验的基础上,进行了分子对接,以说明 QPD-OTf 和驱动蛋白-1的结合。
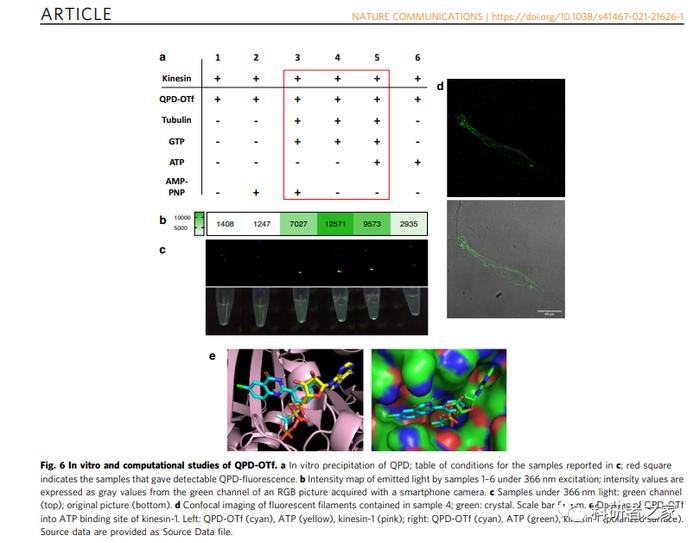
再比如我们平常生信分析里经常会分析到蛋白相互作用,针对某个靶蛋白筛选出一些小分子呀,都可以加上了。实验的时候发现上下游蛋白直接作用.....
分子对接(Molecular docking)就是从原子水平,通过计算机模拟技术,研究分子之间的相互作用过程。
这个工具支持零基础,零代码,基于靶蛋白的小分子、多肽、蛋白对接分析,高清结果图可在论文中直接使用。
比如可以做文献中类似这样的图:
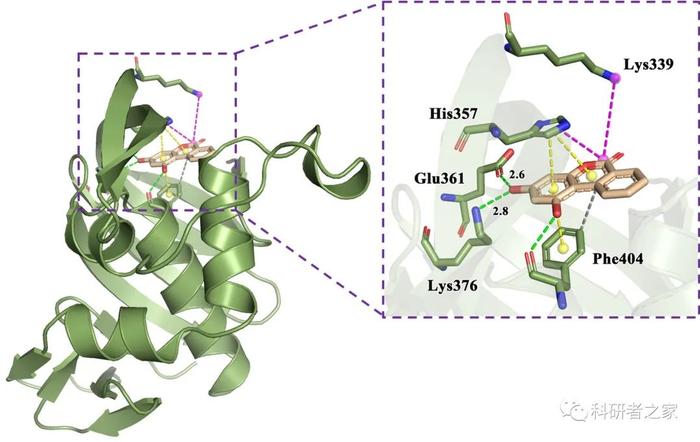
比如
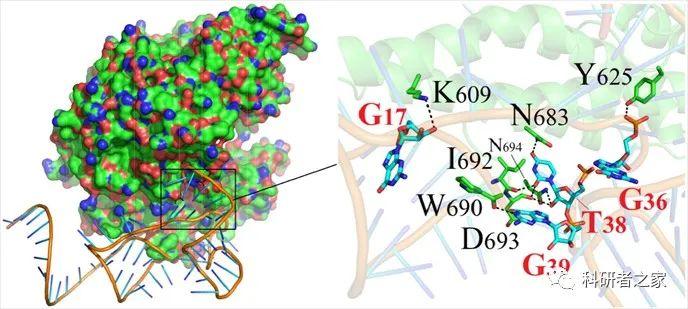
再比如
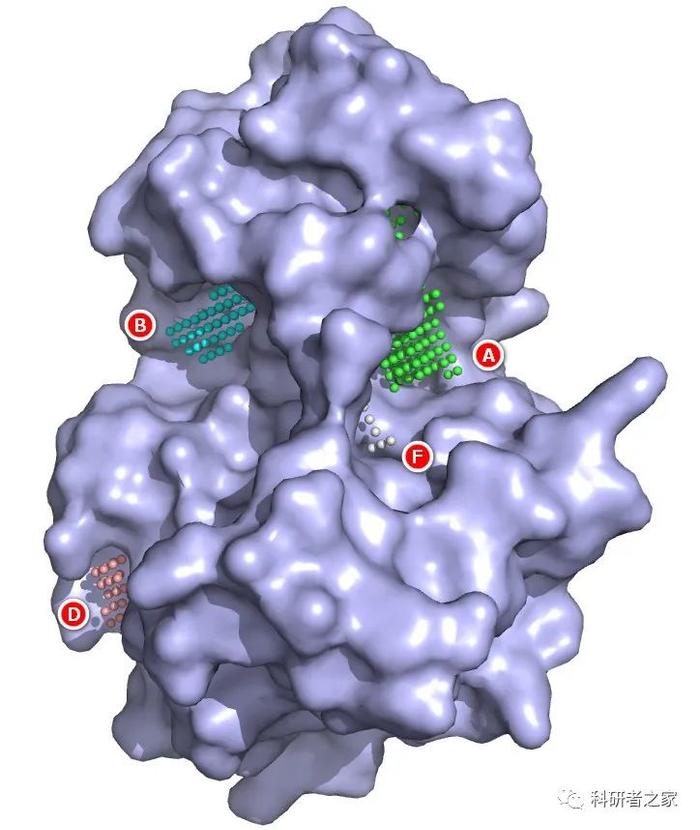
使用方法小白化。
第一步:登录工具,页面简洁无比,顶部支持小分子,多肽,蛋白质和蛋白质的对接分析,分子哪个选哪个。
左边可见只需三步完成:第一步是从PDB数据库选择蛋白的三维结构(PDB是目前收录蛋白三位结果的数据库,基本上你想研究的蛋白都能找到。),可以直接搜关键词或者输入PDB编号。
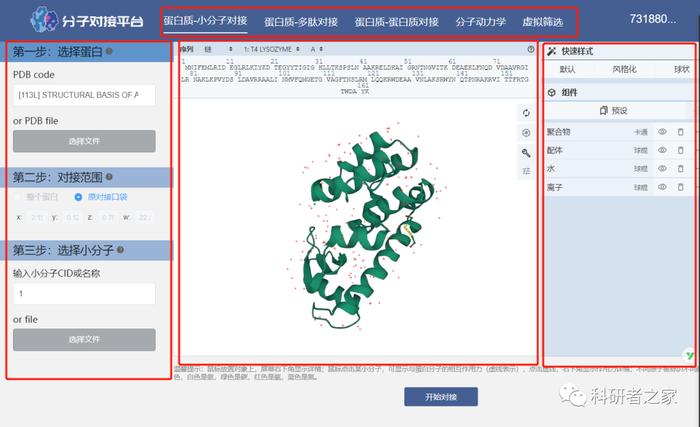
第二步:系统自动会选择使用原对接口袋(位置)还是全蛋白对接范围。
第三步:选择小分子,是从PubChem数据库(也是一个收录小分子化合物的权威数据库,基本都能找到),也可以直接搜关键词或者输入CID编号。
第四步:点击开始对接!
出图!
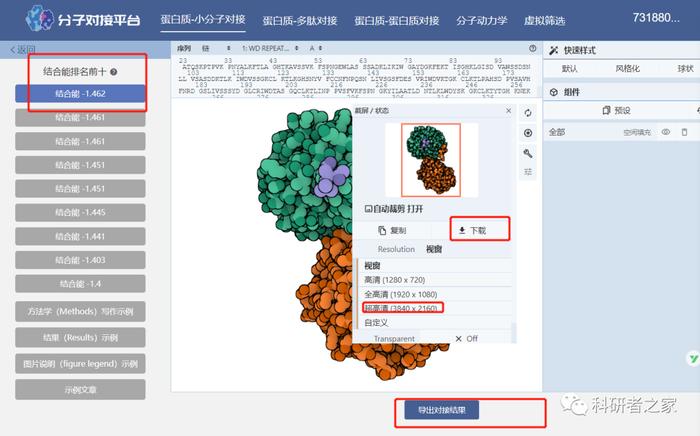
左边按结合能排序,越低越好(好理解,需要结合的能量越低,就越说明能结合)
可以导出对接结果的原始文件,投稿不用怕了。
直接点击下载按钮,设置超高清分辨率,获得高清图片。
文章写作呢?
不怕,方法学,结果,图例写作示例也都有了啦,中英文对照哦,。

收工!可以很自豪的和老板汇报你的学习成果了
老板准夸你:给课题组省下不少钱,Well Done